CITA ESTE TRABAJO
Diego Martínez R, Cano de la Cruz JD, Sánchez Sánchez MI, Vázquez Pedreño LA, Jiménez Pérez M. Desarrollo y entrenamiento de una red neuronal convolucional para la detección de esofagitis en imágenes endoscopicas. RAPD 2025;48(2):48-53. DOI: 10.37352/2025483.1
Introducción
La endoscopia digestiva permite ofrecer una evaluación directa y mínimamente invasiva del tracto gastrointestinal, la evaluación, sin embargo, sigue estando sujeta a cierta variabilidad entre operadores, lo que provoca diferencias significativas en la eficiencia de la técnica según quién la lleve a cabo.
En este contexto, la inteligencia artificial (IA) y, en particular, las redes neuronales convolucionales (CNN, por sus siglas en inglés), han emergido como herramientas prometedoras para mejorar la precisión y reproducibilidad del diagnóstico endoscópico. Estas arquitecturas de aprendizaje profundo, inspiradas en la forma en que las neuronas del cerebro humano se interconectan, están compuestas por múltiples capas de neuronas artificiales que se van entrenando para reconocer patrones a partir de datos. Durante el entrenamiento, cada capa extrae características o rasgos de nivel creciente de complejidad, ajustando constantemente los pesos de sus conexiones a fin de mejorar su capacidad de clasificación o detección. En la práctica clínica actual, la IA se ha comenzado a utilizar en la detección de pólipos colorrectales, la caracterización de lesiones gástricas y otras aplicaciones que buscan apoyar el diagnóstico endoscópico[1],[2].
El presente trabajo se centra en el desarrollo de una red neuronal convolucional entrenada específicamente para la detección de esofagitis en imágenes endoscópicas. A través del uso de técnicas de procesamiento de imágenes y aprendizaje profundo, se busca mejorar la capacidad diagnóstica automatizada. Este enfoque no solo tiene el potencial de mejorar la toma de decisiones clínicas, sino también de agilizar el flujo de trabajo en entornos médicos, facilitando un diagnóstico más rápido y preciso para los pacientes.
En este artículo, se describirá el proceso de desarrollo y entrenamiento del modelo de IA, así como su validación mediante un conjunto de datos de imágenes endoscópicas. Además, se discutirán los desafíos y perspectivas futuras en la integración de estos sistemas en la práctica clínica, con el objetivo de mejorar la calidad del diagnóstico endoscópico y la atención a los pacientes con enfermedades esofágicas.
Materia y Métodos
En este proyecto se ha utilizado el lenguaje de programación Python junto con las bibliotecas Keras y TensorFlow[3] para implementar y entrenar una arquitectura de red neuronal profunda. El entorno de trabajo seleccionado fue Google Colab Pro, que proporciona acceso a potentes unidades de procesamiento gráfico (GPU), en este caso una Nvidia A100. Esto resulta fundamental para reducir significativamente los tiempos de entrenamiento y poder manejar grandes volúmenes de datos de imágenes.
La arquitectura base escogida fue InceptionResNetV2[4], desarrollada originalmente por investigadores de Google y entrenada con el conjunto de datos masivo y público de ImageNet[5]. InceptionResNetV2 combina las ventajas de las convoluciones de la familia Inception con la estabilidad y eficiencia de las conexiones de tipo residual, dando como resultado un modelo que mantiene un equilibrio adecuado entre precisión y velocidad de entrenamiento.
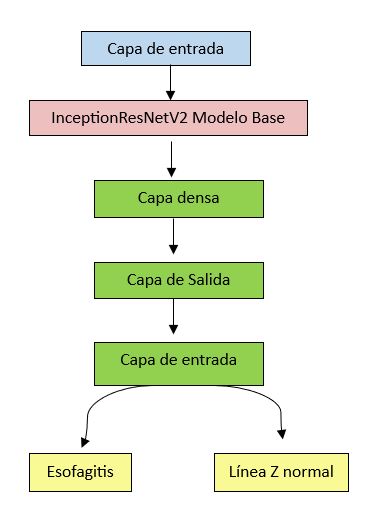
El modelo original de InceptionResNetV2, tras haberse entrenado en la clasificación de miles de categorías de ImageNet, se adaptó para nuestro caso de uso específico. Para ello, se reemplazaron las capas de salida por capas personalizadas diseñadas para realizar clasificación binaria. En particular, se añadieron tres capas densas (completamente conectadas) que culminan en una capa de salida con la activación idónea (para distinguir entre dos clases: esofagitis versus una línea Z normal. (véase Figura 1).
Figura 1
Esquema del modelo creado, en verde las capas añadidas al modelo base InceptionResNetV2 que se muestra en rojo.
El proceso de ajuste fino (fine-tuning en inglés) se llevó a cabo manteniendo fijas las capas iniciales del modelo –las responsables de extraer características– y reentrenando las capas finales específicas. De esta forma, aprovechamos la riqueza de los pesos obtenidos de ImageNet y orientamos la red hacia la discriminación de nuestras dos categorías clínicas de interés. Esto permite un uso mucho más eficiente de los datos y del tiempo de entrenamiento, al evitar entrenar el modelo desde cero.
Entrenamos el modelo con el conjunto de imágenes endoscópicas KVASIR[6], un repositorio que abarca imágenes de endoscopia digestiva. En particular, utilizamos 2000 imágenes correspondientes tanto a la línea Z normal como a diferentes grados de esofagitis, dividiendo los datos en un 80% para entrenamiento y un 20% para validación. Este balance en la partición de los datos permitió una formación robusta del modelo y una evaluación preliminar fiable de su desempeño.
Para llevar a cabo una validación más exhaustiva, empleamos el conjunto de imágenes HyperKVASIR[7], que suma en total 1164; 932 de línea Z normal y 232 de esofagitis. En esta fase, se excluyeron los casos más leves, centrándonos únicamente en los grados B, C y D de la clasificación de Los Ángeles, con el fin de evaluar la capacidad del modelo para identificar lesiones más avanzadas. Adicionalmente, se incorporó un tercer conjunto de imágenes procedentes del Hospital Regional Universitario de Málaga. Este conjunto constaba de 203 imágenes de esofagitis (todas ellas patológicas) que incluían distintas gradaciones de severidad (76 imágenes de grado A de los Ángeles, 42 de grado B, 28 del grado C, 22 del grado D y 18 en la categoría de otros destinada a cuando el endoscopista no especificaba el grado de esofagitis) reforzando así la diversidad y la representatividad clínica de los datos utilizados en el estudio (véase Tabla 1).
Tabla 1
Tabla donde se resumen los conjuntos de datos usados en el proyecto.
Resultados
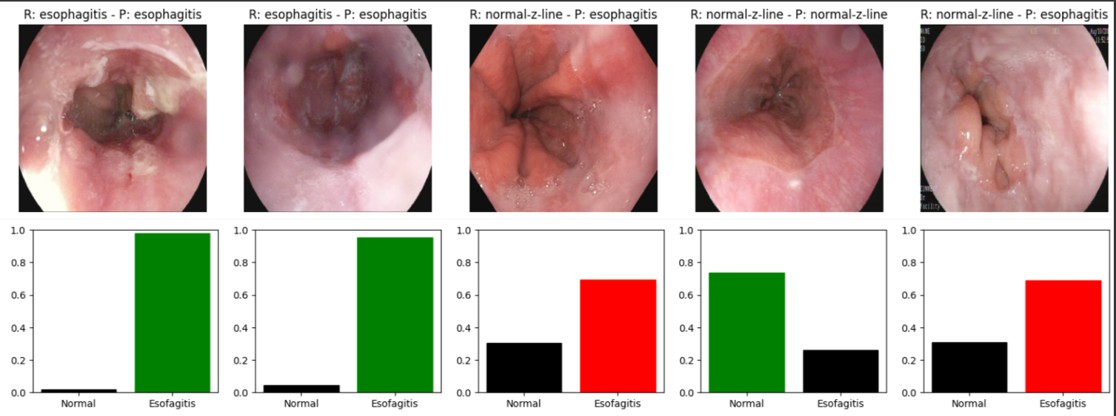
A continuación, se presenta un ejemplo detallado de la capacidad de predicción individual de nuestro modelo, mostrando los porcentajes de confianza asignados a cada categoría diagnóstica. Para ilustrar este aspecto, hemos seleccionado de manera aleatoria cinco imágenes. (veáse Figura 2).
Figura 2
Representación de las predicciones individuales que realiza nuestro modelo. Arriba de cada imágen se muestra la etiqueta real (R:) y la etiqueta predicha (P:) y abajo un gráfico de barras con el porcentaje de confianza que asigna a cada clase en la predicción la cual se tiñe de verde si acierta y rojo si falla.
Al evaluar la detección en los conjuntos de imágenes, se observó lo siguiente. En el conjunto KVASIR—correspondiente al 20% de imágenes reservado para evaluación y no utilizado durante el entrenamiento—de las 200 imágenes que correspondían a línea Z normal, el modelo identificó correctamente 164, mientras que 36 fueron clasificadas erróneamente como esofagitis. De igual forma, de las 200 imágenes que realmente correspondían a esofagitis, 153 fueron correctamente clasificadas, y 47 se confundieron con línea Z normal (véase Tabla 2).
Tabla 2
Matriz de confusión de la predicción de las 400 imágenes del conjunto de imágens KVASIR que corresponden al 20% de imágenes que hemos reservado para la evaluación.
| KVASIR | Línea Z Normal (Predicho) | Esofagitis (Predicho) |
| Línea Z Normal (Real) | 164 | 36 |
| Esofagitis (Real) | 47 | 153 |
Por otro lado, en el conjunto de imágenes HyperKVASIR, la matriz de confusión reveló que, de 932 imágenes de línea Z normal, el modelo clasificó correctamente 833 y falló en 99 casos. Por otro lado, de las 232 imágenes correspondientes a esofagitis, 216 se identificaron correctamente, mientras que 16 fueron erróneamente catalogadas como línea Z normal (véase Tabla 3).
Tabla 3
Matriz de confusión de la predicción de las 400 imágenes del conjunto de imágens HyperKVASIR.
| HyperKVASIR | Línea Z Normal (Predicho) | Esofagitis (Predicho) |
| Línea Z Normal (Real) | 833 | 99 |
| Esofagitis (Real) | 16 | 216 |
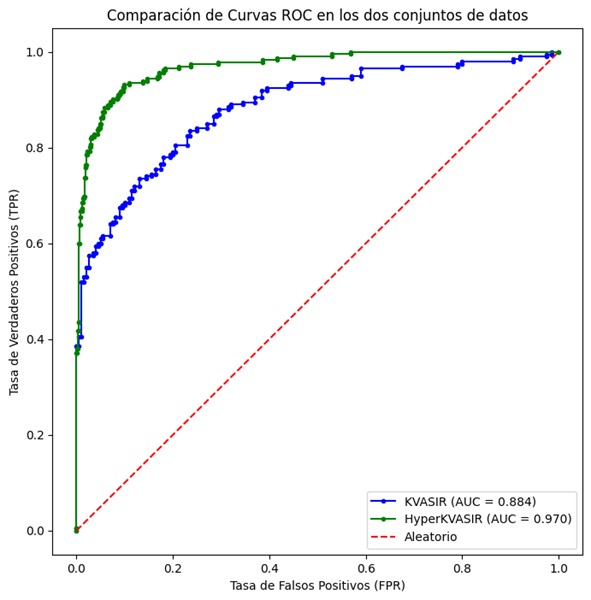
Para comprarar ambas evaluaciones usaremos la métrica de la curva ROC, que es esencial para determinar el desempeño global de nuestro sistema de clasificación. Un aspecto relevante fue la exclusión de esofagitis Grado A únicamente en la base de datos HyperKVASIR, con el fin de evidenciar que al contar con un mayor contraste entre la imagen de control y la imagen patológica, el modelo puede discriminar con mayor eficacia, lo que favoreció la eficiencia en la detección de la patología, logrando valores de área bajo la curva (AUC) de 0.884 para el conjunto de datos KVASIR y 0.970 para el conjunto de datos HyperKVASIR. Estos resultados apuntan a un alto nivel de exactitud diagnóstica. (véase Figura 3).
Figura 3
Representación de las curvas ROC en la validación con el KVASIR (que incluye esofagitis de todo grado de severidad) y HyperKVASIR que solo incluye grados B, C y D descartando los casos más leves) en el que se observa una mejor curva en este último conjunto.
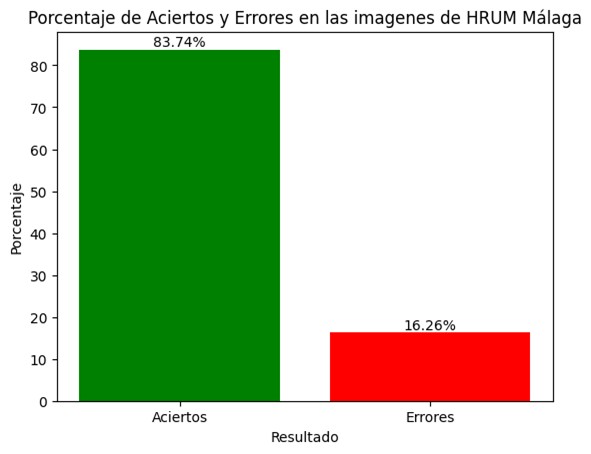
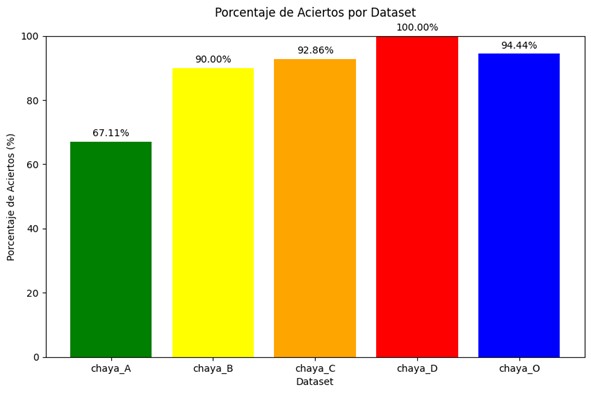
Asimismo, el gráfico siguiente ilustra el porcentaje de aciertos obtenidos por el modelo al ser evaluado con la cohorte del Hospital Regional de Málaga. Este análisis independiente es fundamental para corroborar la aplicabilidad del modelo en entornos clínicos diversos y validar la solidez de la metodología en circunstancias reales. (véase Figura 4).
Figura 4
Porcentaje de aciertos en las imágenes del conjunto creado con casos del Hospital Regional Universitario de Málaga.
Por último, al clasificar el rendimiento según la severidad de la esofagitis, se observó una correlación positiva entre el incremento en la severidad y el porcentaje de aciertos. Este hallazgo sugiere que el algoritmo resulta particularmente eficiente al detectar lesiones más avanzadas. (véase Figura 5).
Discusión
Los hallazgos de este estudio ponen de manifiesto el potencial de la arquitectura InceptionResNetV2 para la detección y clasificación de esofagitis en base a imágenes endoscópicas. El uso de capas preentrenadas con el extenso conjunto de datos ImageNet, junto con el ajuste fino enfocado en el problema de esofagitis vs. línea Z normal.
El valor de la AUC (área bajo la curva ROC) obtenido en los dos principales conjuntos de validación —KVASIR e HyperKVASIR— permitió evidenciar tanto la consistencia como la capacidad de generalización del modelo. La exclusión de grados más leves de esofagitis (grado A) en HyperKVASIR mostró cómo un mayor contraste entre las imágenes normales y las patológicas facilita una discriminación más nítida, reforzando la hipótesis de que el modelo funciona de forma especialmente robusta en lesiones más severas. Este aspecto adquiere relevancia clínica, dado que, en la práctica, las lesiones avanzadas suelen requerir un diagnóstico y tratamiento más oportunos.
El análisis independiente en el conjunto de imágenes del Hospital Regional Universitario de Málaga aporta evidencia adicional sobre la aplicabilidad de la propuesta en entornos diversos. Los resultados confirman la utilidad de la metodología no solo en bases de datos públicas, sino también en un contexto clínico real, con variaciones en las condiciones de captura de la imagen, tipos de equipos endoscópicos y características poblacionales.
El hecho de que el rendimiento del modelo aumente en relación con el grado de severidad de la esofagitis sugiere que la red neuronal es capaz de detectar con mayor precisión las alteraciones estructurales más evidentes. Sin embargo, se hace necesario profundizar en la clasificación de lesiones incipientes, ya que una identificación temprana resulta esencial en la práctica médica para prevenir complicaciones futuras y mejorar el pronóstico de la condición.
A pesar de los resultados prometedores, este estudio presenta algunas limitaciones. Por un lado, el número total de imágenes, aunque significativo, podría ampliarse para abarcar una mayor representatividad de las diferentes formas de presentación de la esofagitis, en especial las de grado A. Por otro lado, factores como la variabilidad en la calidad de la imagen y la presencia de artefactos durante la endoscopia pueden influir en la exactitud del modelo.
Conclusiones
Aunque la utilidad de este modelo resulta todavía poco aplicable a la práctica clínica, este estudio pone en evidencia el potencial de las redes neuronales profundas en la endoscopia y subraya la importancia de la colaboración entre hospitales para crear bases de datos multicéntricas. Aumentar tanto la cantidad como la diversidad de imágenes es crucial para entrenar modelos que, en el futuro, puedan implementarse en contextos clínicos de mayor relevancia. Los resultados aquí obtenidos resaltan que, si bien el modelo muestra alta sensibilidad en lesiones avanzadas, aún se enfrentan retos en la identificación de estadios incipientes y en la adaptación a diversas condiciones de captura de imágenes. Con vistas a la práctica, la validez multicéntrica, el uso de técnicas de aumento de datos y la integración de estos sistemas en flujos de trabajo clínicos podrían, a largo plazo, favorecer diagnósticos más ágiles y precisos para una variedad de patologías gastrointestinales.

 Descargar número completo
Descargar número completo Download full issue
Download full issue